概要
シングルセル遺伝子発現
Single Cell RNA-seqは複雑な組織や疾患の研究に革命をもたらす解析手法です。Partek Flowに搭載された強力で使いやすい解析ツールによりSingle Cell RNA-seqのデータを解析できます。
 各社のシングルセル技術に対応
各社のシングルセル技術に対応

Partek FlowだけでSingle Cell RNA-seqのデータ処理やデータ解析ができます。Drop-seqや10x Genomicsで得られたFASTQファイルやカウントファイルを読み込むと、Partek Flowは簡単なステップを組み合わせてすべてのデータ処理を行います。各ステップはパラメーターを自由に設定でき、処理内容の詳細をいつでも確認できます。
Single Cell RNA-seqのデータは通常のRNA-seqのデータと比べてより複雑です。それぞれのリードが元となる細胞を標識するバーコードを持ち、逆転写されたそれぞれのcDNAがUMIを持ちます。Partek Flowは参照ゲノム配列にアラインメントされたリード配列からバーコードやUMIを簡単に認識して除去できます。UMIやバーコードによりPCRアーティファクトやノイズを除去できます。
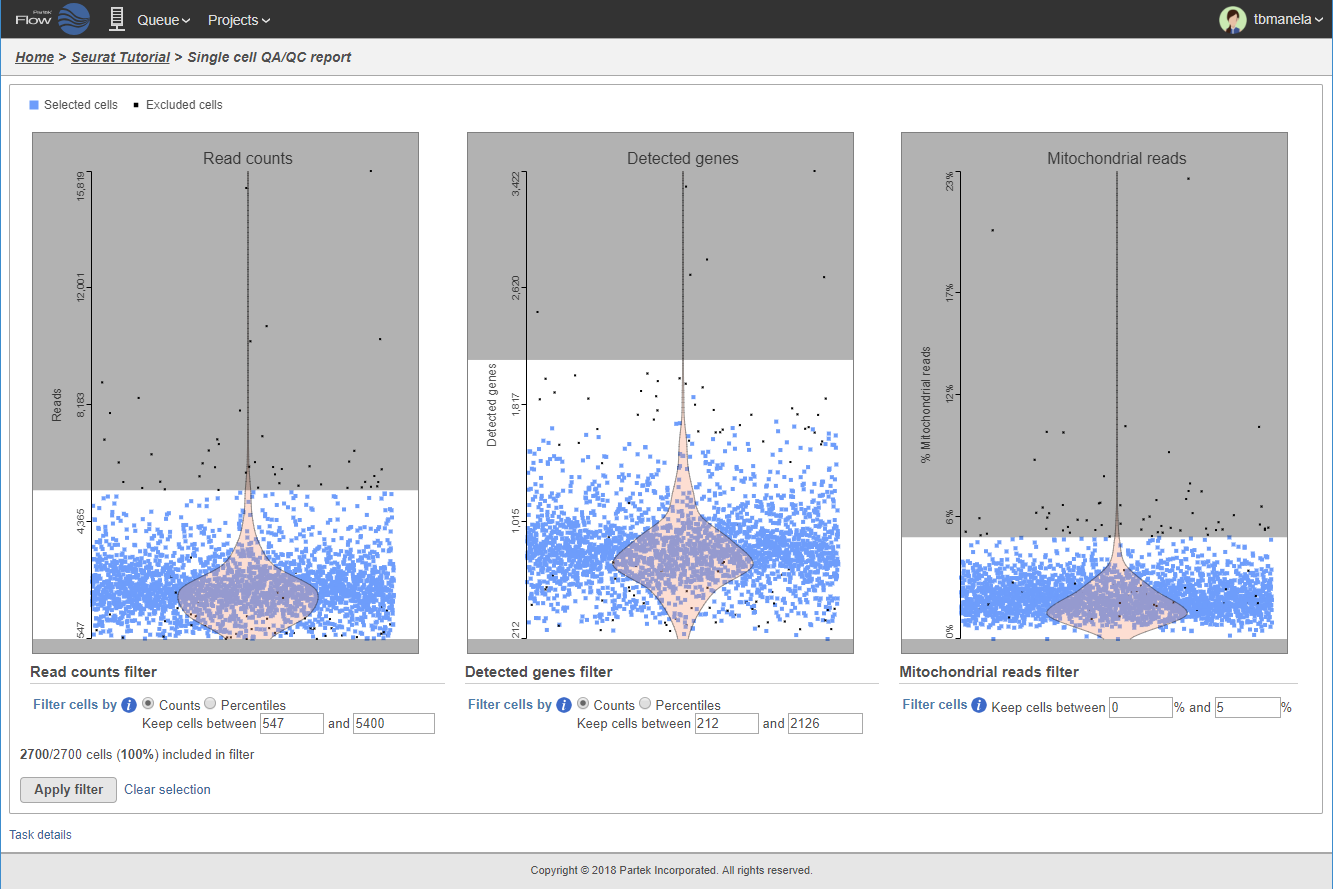
細胞ごとの遺伝子発現データが得られたら、マウス操作で簡単に品質の低い細胞や発現が低い遺伝子を除外できます。細胞当たりのリード数や遺伝子数あるいはミトコンドリア由来リードの割合を指標にして、一細胞になっていない細胞や正常ではない細胞を除外できます。また、発現量や分散あるいは遺伝子IDによってその後の解析を行う遺伝子を絞り込むこともできます。
 シングルセルデータの中で細胞集団の探索
シングルセルデータの中で細胞集団の探索
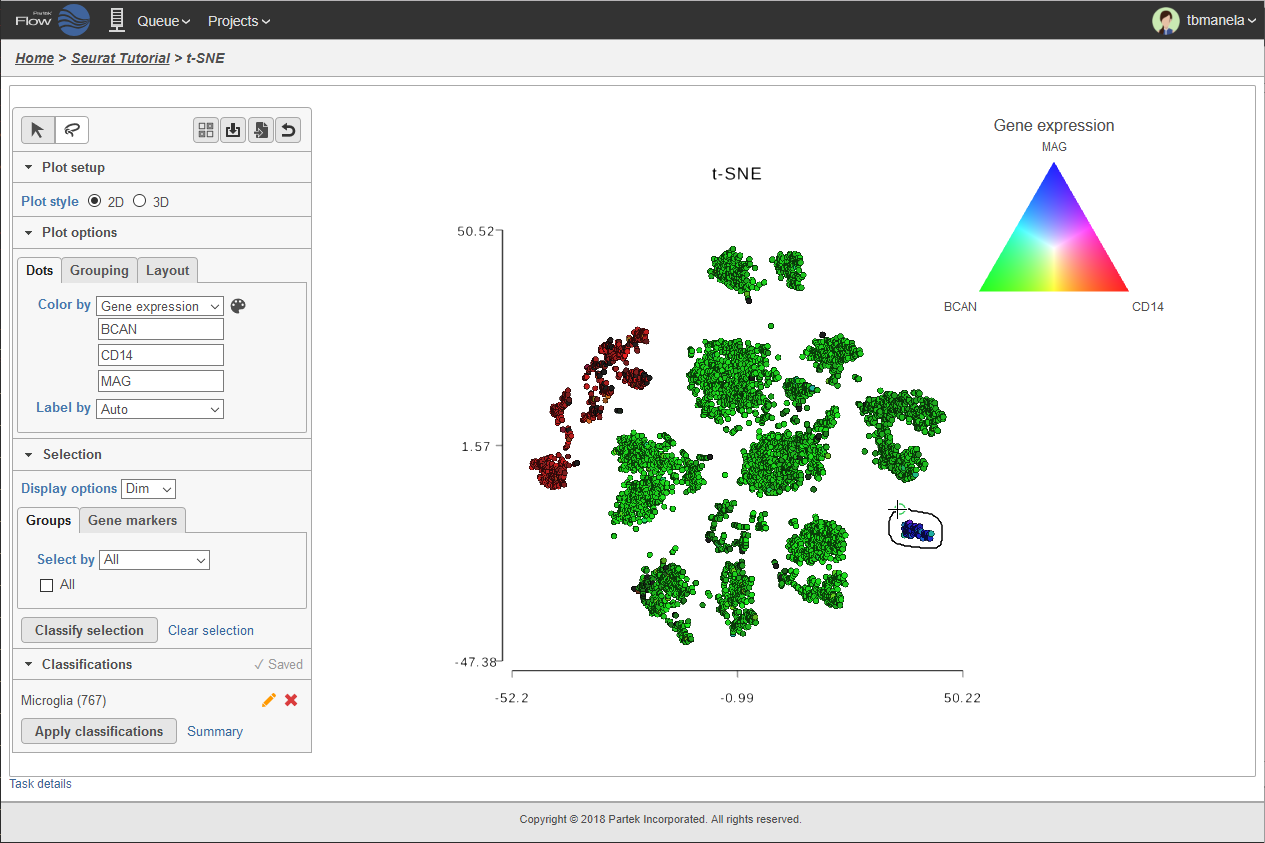
t-SNE (t-distributed stochastic neighbor embedding:t分布型確率的近傍埋め込み)は、スキャッタープロット上に発現パターンが似ている細胞集団をクラスターとして表示する次元削減のアルゴリズムです。Partek Flowでは、インタラクティブなt-SNEプロットはシングルセルデータの中で細胞集団を探索するための強力なツールです。t-SNEプロット上に遺伝子やパスウェイの発現を重ねて表示することで、既知の細胞集団を同定できます。未知の細胞集団に対しては、Partek Flowで利用可能なクラスタリング手法により発現パターンが似ている細胞集団を特定でき、それぞれのクラスターに対するバイオマーカーを検出して細胞集団の同定を迅速化できます。
細胞集団の同定に当たって、Partek Flowに搭載された強力で自由度の高い統計検定により細胞集団間あるいは細胞集団の異なる実験条件間で発現が変動する遺伝子やパスウェイを検出できます。サンプル間の細胞集団の比率を比較することで、組織における細胞集団の構成を変化させる実験条件を識別することもできます。
 シングルセル解析結果の表示
シングルセル解析結果の表示

Partek Flowでのすべての解析結果はカスタマイズ可能な可視化ツールで表示できるので、シングルセル解析の結果を簡単に表示したり共有したりできます。
異なる細胞集団間での発現パターンを可視化したりクラスタリング結果を表示する2次元あるいは3次元のt-SNEプロットは画像としてダウンロードできます。
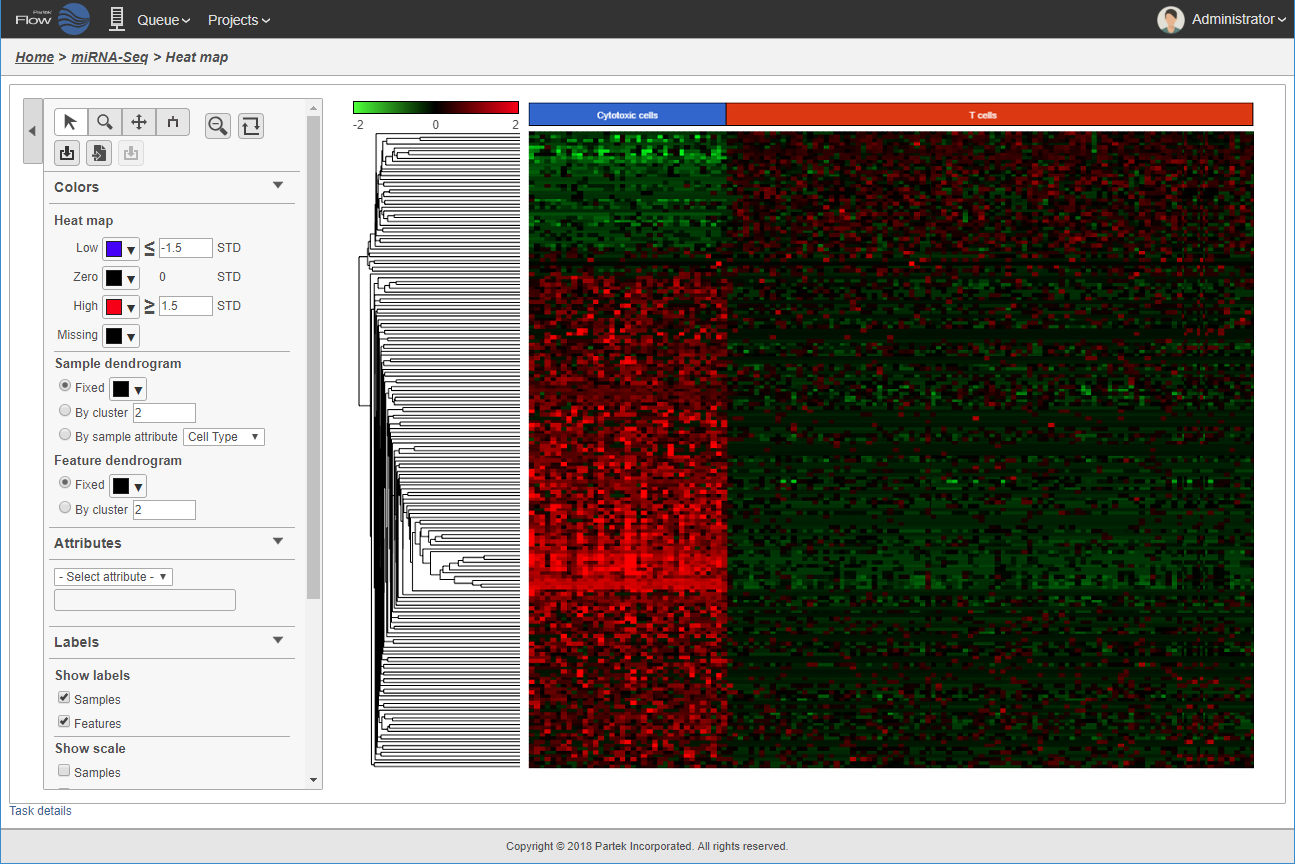
カスタマイズ可能な階層型クラスタリングのヒートマップやバイオリンプロットも設定したサイズや解像度の画像してダウンロードできます。
遺伝子発現変動は遺伝子ごとにグラフ表示したり、パスウェイごとにKEGGパスウェイマップ上に重ねて表示したりできます。


